本程式教學使用primates.nex當作範例。
Quick Start Version(懶人版)
要用MrBayes分析類緣關係有四個步驟:
1. 讀取Nexus檔案(Read the Nexus data file)
2. 設定演化模型(Set the evolutionary model)
3. 跑分析(Run the analysis)
4. 對樣本數進行摘要擷取(Summarize the samples)
1. 在MrBayes>命令提示字元後面,輸入execute primates.nex。這個動作會把資料帶進程式。不過請留意MrBayes會假設你把資料檔案(primates.nex)放在和MrBayes程式同一個資料夾,如果你的資料檔案放在其他地方,請輸入資料檔案的路徑,例如 execute ../taxa/primates.nex。不過建議把資料檔案放在MrBayes檔案夾裡面,比較不會有問題。如果使用自己的資料,而且資料裡面已經有一些MrBayes程式的指令,那可能會影響會改變這個懶人版程式教學的執行過程,所以可以先刪除那些指令或是把那些指令放在[]中括號裡面。
2. 在MrBayes>命令提示字元後面,輸入lset nst=6 rates=invgamma。這是為了要設定演化模型成為GTR模型,而且DNA序列有gamma-distributed rate variation across sites and a proportion of invariable sites。如果你的資料不是DNA或RNA,如果你要選model,或是你要改變其他設定,請看使用手冊。
3.1 在MrBayes>命令提示字元後面,輸入mcmc ngen=10000 samplefreq=10。這樣會讓你至少從事後機率分佈(posterior probability distribution)得到一千個樣本。如果是大型資料,你可能會想要跑更多的分析,而採樣的頻率會減少(程式預設是一百萬個generation,採樣頻率則是百分之一),在這種情況下,你可以預測分析大概所需要花的時間是多少。
3.2 如果在10000 generations以後,standard deviation of split frequencies小於0.01,當程式問 "Continue the analysis? (yes/no)"的時候就回答no停止程式,如果大於0.01則繼續增加generations直到小於0.01。
4.1 輸入sump burnin=250來對參數值進行摘要擷取(250或是25%的generations)。程式會自己輸出樣本的鹼基取代模型參數,包括各種參數的平均值、模式和95%的信賴區間。請確認所有參數的potential scale reduction factor (PSRF)接近1.0,如果沒有的話請跑更長的分析(請看步驟4.2的第一張圖,表格最右邊那一欄就是PSRF)。
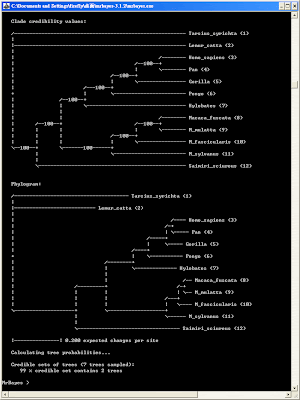
4.2 輸入sumt burnin=250來對演化數進行摘要擷取(250或是25%的generations)。程式會自己輸出一棵每個分支都有事後機率(posterior probabilities)的支序樹(cladogram),以及一顆有平均分支長度(mean branch lengths)的譜系樹(phylogram)。這些樹可以被 TreeView, MacClade, Mesquite等軟體讀取、列印和編輯。






1 則留言:
不好意思,請問你知道cool chain stock 的可能原因嗎?
如何做trouble shotting ?
thanks
Vanson
張貼留言