 請連結到MrBayes Block Form。(New Link)
請連結到MrBayes Block Form。(New Link)在MrBayes懶人版或是完整版裡面,已經有提到各種設定和指令,不過都要一個指令一個指令慢慢輸入,很容易有打錯字、忘記打過什麼、不確定已經輸入的參數對不對的問題,而就有好心人弄了一個網站,做了一個比懶人版還要更懶更方便更好用的方法,滑鼠點幾下,複製貼上就可以了,真好。
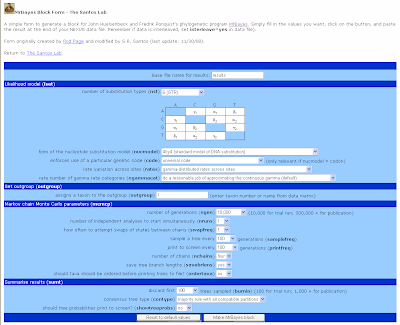
請先連結到上面那個網站,然後按照步驟來:
1. Base file name for results:請在這一格裡面輸入你希望分析結果的檔案名稱是什麼,預設是result,你也可以改成Fish1、Fish2等等的。這樣MrBayes分析完就會輸出Fish1.t、Fish1.con等檔案。
2. number of substitution types (nst):請選擇一個model吧!有三個選項:1 (J69 or F81) 2 (K80 or HKY85) 6 (GTR)
3. form of the nucleotide substitution model (nucmodel) :也有三個選項,詳細解釋請看MrBayes完整版。doublet、4by4 、codon
4. enforces use of a particular genetic code (code):如果nucmodel選擇codon的話,這裡才需要設定,看要分析的資料屬於哪一種。
5. rate variation across sites (rates):有五個選項。
6. rate number of gamma rate categories (ngammacat):有三個選項:正常、較長的分析、更長的分析。
7. assigns a taxon to the outgroup (outgroup):請輸入外群的號碼或名字
8. number of generations (ngen):這個網站建議測試時設定為10000,用來發表請至少設定為500000。
9. number of independent analyses to start simultaneously (nruns):例如完整版的設定為2。
10. how often to attempt swaps of states between chains (swapfreq):例如完整版的設定為1。
11. samplefreq:每幾個generations採樣一顆樹。例如完整版的設定為100。
12. printfreq:幾個generations在螢幕上秀出結果。例如完整版的設定為100。
13. number of chains (nchains) : 例如完整版的設定為four。
14. save tree branch lengths (savebrlens) :如果要儲存分支長度請選yes。
15. should taxa should be ordered before printing trees to file? (ordertaxa) :要不要在輸出樹之前,taxa要先被排序。
16. burnin:這個網站建議測試時設定為100,用來發表請至少設定為1000以上,或是用25%的ngen來設定。
17. consensus tree type (contype):看要用哪一種方法做出consensus tree。
18. should tree probabilities print to screen? (showtreeprobs):一般選no即可。
現在假設我完全按照這個網站的預設值,然後按Make MrBayes block,會得到這些結果:
可以把這些文字複製後,直接貼在要分析的資料下面就可以了。例如原本的資料Nexus檔案為:
#NEXUS
begin data;
dimensions ntax=12 nchar=898;
format datatype=dna interleave=no gap=-;
matrix
:
:
:
end;
貼上後變成:
#NEXUS
begin data;
dimensions ntax=12 nchar=898;
format datatype=dna interleave=no gap=-;
matrix
:
:
:
end;
begin mrbayes;
lset
nst=6
nucmodel=4by4
code=universal
rates=gamma
ngammacat=4;
[ Set the outgroup for the analysis ]
outgroup 1;
[ Set Markov chain Monte Carlo parameters ]
mcmcp
ngen=10000
nruns=1
swapfreq=1
printfreq=100
samplefreq=100
nchains=4
savebrlens=yes
ordertaxa=no
filename=results;
[ Go! ]
mcmc;
[ Summarise the results ]
sumt
filename=results
burnin=100
contype=allcompat
showtreeprobs=no;
end;
然後儲存檔案,再執行就可以,例如exe primates.nex,就可以直接把分析跑完,不必再打其他指令。而如果要改其中的參數,也不必回到這個網站改完再複製貼上,直接使用文字編輯器打開Nexus檔案改就好,例如把ngen=1000改成ngen=10000,就只要多打一個0就好了。

沒有留言:
張貼留言